植物基因组中的单碱基变异(single-nucleotide variations,SNVs)、多碱基变异(multiple nucleotide variations,MNVs)和结构变异(structural variations ,SVs)对于基因功能研究和生物学性状提升有着起着重要作用。但现有基因组编辑方法受制于编辑精度有限、编辑事件非定向等因素,难以实现上述变异材料的有效定向获得。2019年,David Liu课题组开发的引导编辑器(prime editor, PE)经多次迭代升级,目前在哺乳动物细胞中已实现任意碱基替换编辑及插入删除等结构变异的有效创制,然而植物中引导编辑效率仍然较低,限制了SNVs,MNVs和SVs在重要农作物基因组中的有效获得。

近日,Plant Communications杂志在线发表了西南大学生命科学学院张勇团队题为“An improved plant prime editor for efficient generation of multiple-nucleotides variations and structural variations in rice”的研究论文。该研究针对目前水稻基因组引导编辑效率有限、多类型编辑事件实现困难等问题,基于N-terminus PE+epegRNA的策略,开发了新型引导编辑器NEPE (N-terminus M-MLV RT fused to Cas9 nickase plus epegRNA prime editor),并在水稻中实现了高效的SNVs,MNVs和SVs创制,为水稻基因组精准编辑提供了有效工具。全文主要结果简述如下:
基于蛋白质元件优化,设计构建了4种水稻引导编辑PE2系统(图1A),并在水稻原生质体中对四种PE2系统的编辑效率进行了快速评价。经通量测序分析表明,在所选择已报道的3个位点处,N-PE2系统编辑效率最高,平均编辑效率约为10%(图1B)。而选用epegRNA(pegRNA+ EvopreQ1)可极大地提升编辑效率(图1B-D)。因此,将N-PE2+epegRNA命名为NEPE系统。值得一提的是,在所测试的OsALS1位点处,NEPE系统较此前发表的ePEmax2系统编辑效率更高(图1E)。上述结果表明,引导编辑器融合蛋白结构的简单变化结合epegRNA可以极大地提升水稻中的引导编辑效率。
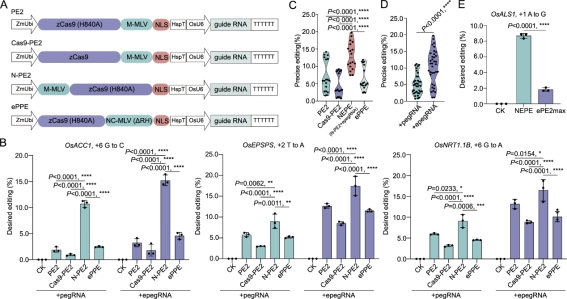
图1 NEPE系统构建及编辑效率评价
基于NEPE系统进一步测试10个水稻内源位点SNVs创制可行性,经水稻原生质体转化及通量测序分析,明确了NEPE在水稻基因组中实现有效碱基替换、插入及删除编辑的可靠性(图2A-D)。在水稻T0代植株中,NEPE系统显示了22.9%~30.0%的编辑效率,其中双等位编辑效率为2.1%~10.0%(图2C-G),实现了终止密码子精准引入的基因敲除(图2D)及抗除草剂基因新位点定向进化(图2E-G),
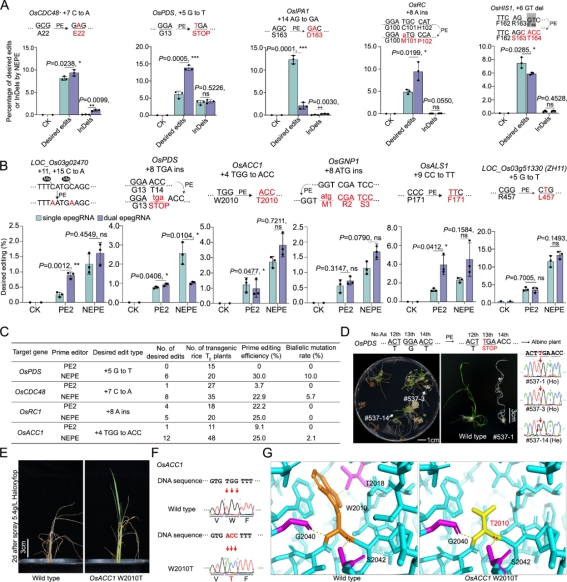
图2 NEPE介导的水稻基因组SNVs创制
进一步,针对水稻OsGRF4中OsmiR396结合位点进行MNVs创制,在同义替换前提下,对7个核苷酸同时进行编辑(图3A),在水稻T0代植株中,NEPE编辑效率最高为21.1%,双等位编辑效率为5.1%(图3B),有效解除了OsmiR396对OsGRF4的表达量调控,最终通过提升OsGRF4表达量提升农艺性状(图3C-J)。
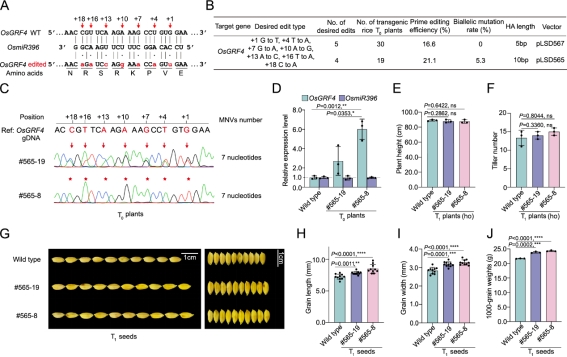
图3 NEPE介导的水稻基因组MNVs创制
进一步,作者使用NEPE系统对水稻OsWx基因启动子区域进行了位点设计(图4A),在水稻原生质体、T0代植株中实现了98bp~523bp的精准片段删除,NEPE系统的删除效率、精准度较PE2、Cas9-PE2系统更高(图4B-E)。随后,作者应用NEPE系统,在OsMSP基因中实现了FLAG tag、His tag的有效插入,编辑效率为15.0%~20.0%,双等位编辑效率为4.0%~5.0%,并经western blotting明确了插入事件的可靠发生(图4F-J)。最后,作者结合精准删除及插入,针对蛋白质内源区域实现了结构域替换,同时实现了3900bp删除及34bp插入,精准替换效率为45.0%(图4K-L)。
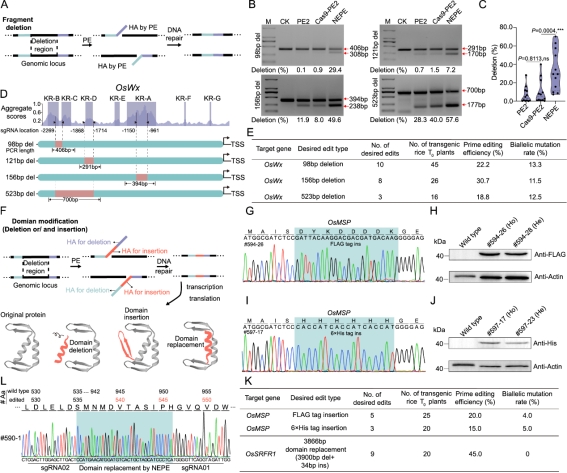
图4 NEPE介导的水稻蛋白质编码基因启动子及氨基酸序列SVs事件创制
电子科技大学博士后仲昭辉(现为四川农业大学水稻研究所特聘副教授)、博士研究生范婷婷为论文共同第一作者,西南大学生命科学学院张勇教授为论文通讯作者,研究工作得到STI-2030重大项目、国家自然科学基金面上及青年项目资助。
论文链接:https://www.cell.com/plant-communications/fulltext/S2590-3462(24)00284-0